Chandra/HETGの位相分割スペクトル抽出の解析ログ
誰かの参考になるかと思い、Chandra/HETGのphase-resolved spectral extractionをしたときのログを掲載しておきます。 V1223 Sgrという白色矮星のスペクトルをスピン周期で畳み込んだスペクトルを抽出し、プロットしています。 生成される結果は、以下のようなPDFファイルです。
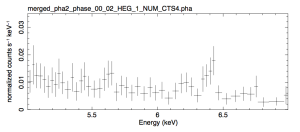 この例では、周期を5分割してスペクトルを抽出していますが、exposureは元の値が入ったままになっているので、時間平均スペクトルと比べて縦軸が1/5になります。適当にスケールしてください。
この例では、周期を5分割してスペクトルを抽出していますが、exposureは元の値が入ったままになっているので、時間平均スペクトルと比べて縦軸が1/5になります。適当にスケールしてください。
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
#---------------------------------------------
# Barycentric correction
#---------------------------------------------
axbary infile=acisf00649_repro_evt2.fits orbitfile=../primary/orbitf073397100N001_eph1.fits outfile=acisf00649_repro_evt2_bary.fits rad=283.759625 dec=-31.163778
#---------------------------------------------
# Put phase column
#---------------------------------------------
punlearn dmtcalc
dmtcalc infile=acisf00649_repro_evt2_bary.fits outfile=acisf00649_repro_evt2_bary_phase.fits expression=@dmtcalc.expression
#---------------------------------------------
# Extract
#---------------------------------------------
function extractPhaseResolvedSpectrum(){
dmcopy "acisf00649_repro_evt2_bary_phase.fits[phase=${phaseFilter}]" ${eventFile} clobber=yes
dmappend infile="acisf00649_repro_evt2.fits[REGION]" outfile=${eventFile}
tgextract infile=${eventFile} outfile=${phaFile} clobber=yes << EOF
EOF
}
phaseFilter=0.0:0.2
eventFile=evt2_phase_00_02.fits
phaFile=pha2_phase_00_02.fits
extractPhaseResolvedSpectrum
phaseFilter=0.2:0.4
eventFile=evt2_phase_02_04.fits
phaFile=pha2_phase_02_04.fits
extractPhaseResolvedSpectrum
phaseFilter=0.4:0.6
eventFile=evt2_phase_04_06.fits
phaFile=pha2_phase_04_06.fits
extractPhaseResolvedSpectrum
phaseFilter=0.6:0.8
eventFile=evt2_phase_06_08.fits
phaFile=pha2_phase_06_08.fits
extractPhaseResolvedSpectrum
phaseFilter=0.8:0.999
eventFile=evt2_phase_08_10.fits
phaFile=pha2_phase_08_10.fits
extractPhaseResolvedSpectrum
#---------------------------------------------
# Merge
#---------------------------------------------
n=1
for eventFile in `ls pha2*fits`; do
add_grating_orders
pha2=${eventFile} order=${n} garm=HEG garfm=tg/acisf00649_repro_heg_m${n}.arf garfp=tg/acisf00649_repro_heg_p${n}.arf
root=merged_`basename ${eventFile} .fits` gtype=NUM_CTS gspec=4 verbose=2 clobber=yes
done
#---------------------------------------------
# Plot
#---------------------------------------------
for file in `ls merged_pha2_phase_*CTS4.pha`; do
xspec << EOF
data $file
response tg/acisf00649_repro_heg_m1.rmf
cpd /xs
ignore **-5.5 7.5-**
plot data
setplot rebin 3,3,1
plot
iplot
log x off
log y off
r y 0 0.035
ti off
view 0.1 0.1 0.9 0.5
la t
la f $file
lab 2 pos 6.4 0 line 90 1 col 8 ls 1 "
!"
plot
hard `basename $file .pha`.ps/cps
exit
exit
EOF
ps2pdf `basename $file .pha`.ps
done